Desde la perspectiva humana, los hongos son Sol y sombra: pueden ser los proveedores de una gran diversidad de metabolitos y materiales útiles para la industria alimentaria y biomédica, y al mismo tiempo pueden ser patógenos de humanos y plantas a los que hay que combatir.
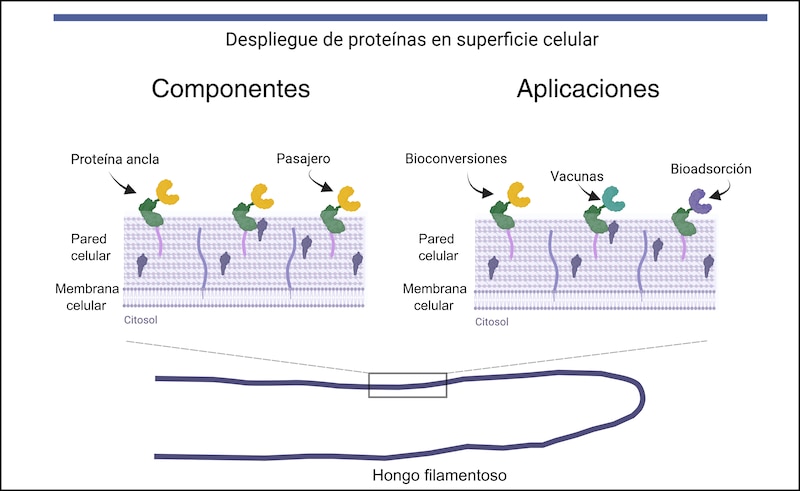
Tanto para explotarlos como para controlarlos, es indispensable conocer mejor estos microorganismos. Una de las áreas de investigación más activas en hongos es la de la síntesis, ensamblaje y dinámica de su pared celular. La pared celular es una coraza rígida que cubre a las células fúngicas (Figura 1). Esta coraza es la que interactúa con el medio ambiente y todos sus agentes estresantes; además, su síntesis y ensamblaje también dirigen el crecimiento y la forma de los hongos. Entender el origen y dinámica de la pared celular de los hongos no solamente permitiría controlar su crecimiento y propagación, muy benéfico para la medicina y la agricultura, también sería útil para manipular ese crecimiento en fermentaciones para la obtención de bioproductos útiles. Manipular genéticamente a los hongos para decorar su pared celular con proteínas que agreguen o mejoren sus propiedades también traería grandes beneficios en áreas como la biorremediación, la generación de vacunas o la producción consolidada de biocombustibles (Figura 1).
La multifacética pared celular de los hongos: rígida, pero plástica y móvil
Las paredes celulares fúngicas están compuestas de polisacáridos (glucanas, quitina, entre otros), así como proteínas (Figura 1). Al día de hoy, la pared celular de los hongos es vista como un complejo que parece rígido, pero en realidad su composición es dinámica ya que se adapta al ambiente. Además, recientemente se ha visto que en la pared hay islotes de polisacáridos que han exhibido una inesperada movilidad molecular (Latgé y Wang, 2022). La rigidez-plasticidad-movilidad de la pared celular fúngica debe ser modulada muy finamente para que, al mismo tiempo que provee protección mecánica a la célula, no impida su crecimiento. No obstante todo el conocimiento acumulado sobre los polisacáridos que forman la pared celular, la investigación sobre las proteínas que residen en ella había quedado relegada, y aunque se conocía que algunas de ellas son enzimas que participan en la remodelación y maduración de los polisacáridos de la pared, la mayoría de ellas eran clasificadas, no sin desdén, como proteínas “estructurales” poco relevantes por carecer de actividad enzimática.
Las atípicas proteínas de la pared celular
Sin embargo, las características de las proteínas de la pared celular fúngica sugieren una especialización funcional, además de mostrar un patrón de evolución único. A diferencia de las proteínas del interior de la célula, es difícil detectar ortólogos (proteínas de diferentes especies que provienen del mismo gen ancestral) de una proteína de pared más allá de especies filogenéticamente próximas (Urbar-Ulloa J, Montaño-Silva P, Ramírez-Pelayo S et al, 2019). Esto ha sugerido que las proteínas de la pared sufren mutaciones a un ritmo más acelerado que las del interior celular, haciendo posible la generación de una gran diversidad proteica en la superficie del hongo, lo que, eventualmente, le permitiría adaptarse a medios cambiantes.
Las proteínas de la pared celular más abundantes son las proteínas GPI, llamadas así porque son modificadas postraduccionalmente (después de que son sintetizadas en el ribosoma) con un glicolípido (glicosilfosfatidilinositol) que les permite llegar a la membrana plasmática y, una vez allí, ser enlazadas covalentemente con la pared celular. El segundo grupo en abundancia son las proteínas con repeticiones internas, comúnmente llamadas PIR por sus siglas en inglés (Figura 2). Las proteínas PIR se caracterizan por contener secuencias definidas (motivos PIR) que se repiten de 1 a 12 veces dentro de la misma proteína. En levaduras como las que sirven para la producción de cerveza, los motivos PIR se repiten uno tras otro cerca del inicio de la proteína (extremo N-terminal), aunque también contienen un dominio rico en cisteínas en el final de la misma, el C-terminal. Tanto los motivos PIR como el dominio rico en cisteínas se unen covalentemente a los polisacáridos de la pared y, en levaduras, se han relacionado con su estabilidad (Urbar-Ulloa J, Montaño-Silva P, Ramírez-Pelayo S et al, 2019). Debido a la unión bimodal de las proteínas PIR con la pared celular, se les ha usado muy frecuentemente como anclas moleculares para el despliegue de proteínas en la superficie de levaduras (Figura 1), lo que aumenta sus propiedades biotecnológicas.
Las proteínas PIR sólo son codificadas por ascomicetos, pero en hongos filamentosos su arquitectura está invertida
Con la intención de extender la utilidad biotecnológica de las proteínas PIR a los hongos filamentosos, recientemente se investigó la diversidad y funcionalidad de las proteínas PIR en el reino Fungi, lo que condujo a hallazgos inesperados (Montaño-Silva, P. et al, 2025). Sorpresivamente, sólo se encontraron proteínas con motivos PIR entre los ascomicetos, uno de los dos tipos más abundantes de hongos, por lo que es posible que las proteínas PIR hayan sido una innovación para resolver un reto evolutivo particular de estos microorganismos. Además, las proteínas PIR fueron más numerosas y con mayor número de dominios PIR entre las clases de hongos que no habitan un nicho ecológico definido y que requieren de mecanismos flexibles de adaptación a medios ambientes diversos, lo que sugiere que las proteínas PIR son parte de ese mecanismo de adaptación.
Entre los hongos filamentosos también se encontraron proteínas PIR; sin embargo, en ellos la arquitectura de las proteínas está invertida, es decir, los motivos PIR se encuentran próximos al extremo C-terminal de la proteína, mientras que el dominio de cisteínas está cerca del extremo N-terminal (Figura 2). A pesar de la inversión de su arquitectura, las proteínas PIR de hongos filamentosos parecen tener la misma función que las de levaduras, es decir, estabilizar la pared celular.
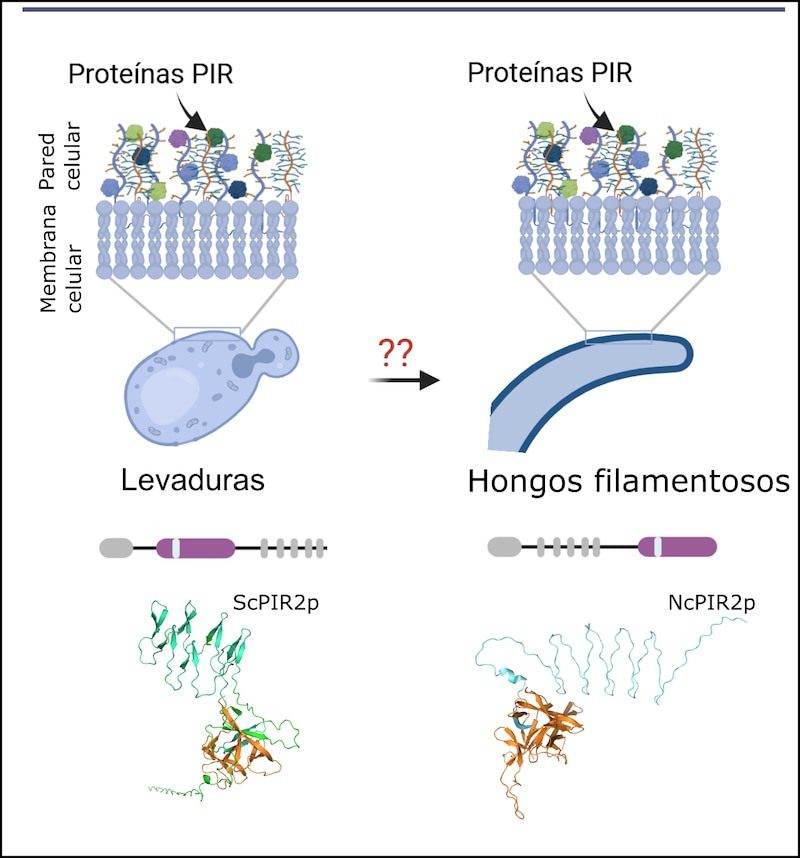
Las proteínas PIR son las segundas más abundantes en la pared celular y tienen la función de estabilizarla bajo un mecanismo que aún se desconoce. Las proteínas PIR de levaduras (izquierda) acumulan los dominios PIR (bloque morado) cerca del extremo N-terminal de la proteína (el comienzo de la proteína), mientras que el dominio rico en cisteínas (bloquecitos grises) está más próximo al extremo C-terminal (el final de la proteína). En contraste, las proteínas PIR de hongos filamentosos (derecha) tienen una arquitectura invertida: el dominio de cisteínas está más cercano al N-terminal de la proteína, mientras que los dominios PIR están más cercanos al extremo C-terminal. Sin embargo, ambas clases de proteínas PIR tienen la misma función estabilizadora de la pared celular.
Como si lo anterior no fuera suficiente, tanto en las proteínas PIR de levaduras como en las de hongos filamentosos se encontró un motivo PIR de secuencia invertida embebido en el dominio de cisteínas. Todas estas características (variabilidad del número de proteínas PIR dentro de la misma especie de hongo, multiplicación del número de motivos PIR dentro de la misma proteína, inversión de la arquitectura de la proteína PIR, y generación de motivos PIR de secuencia invertida), son mecanismos de generación de diversidad dentro de las proteínas PIR, lo que las confirma como típicas proteínas de pared abocadas a generar la mayor diversidad de secuencia posible que permite al hongo adaptarse a ambientes cambiantes.
Las proteínas PIR de hongos filamentosos no cubren de manera homogénea la pared celular, entonces ¿cómo la estabilizan?
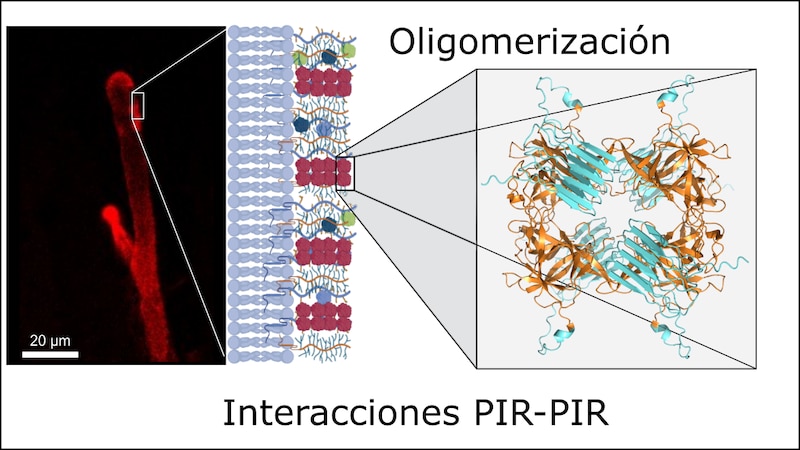
Experimentos de inmunofluorescencia con anticuerpos que identifican específicamente la fusión de la proteína PIR-1 de Neurospora crassa con el epítopo V5, demostraron que las proteínas PIR no cubren homogéneamente la pared celular, sino que se acumulan como parchecitos (izquierda). Hipotetizamos que esos parchecitos son estabilizadores locales de islotes de β-1,3-glucanos, los polisacáridos a los que se unen las proteínas PIR. Además, debido a las regiones intrínsecamente desordenadas de las proteínas PIR, es posible que esos parchecitos estén formados por oligómeros de las proteínas PIR (derecha).
Fusiones de estas proteínas estabilizadoras de la pared celular con una secuencia (V5) que es capaz de unir anticuerpos específicos y con la proteína verde fluorescente (GFP), una proteína que fluoresce cuando se le hace incidir un láser haciéndola visible en un microscopio confocal, mostraron sorpresivamente que las proteínas PIR se acumulan como parchecitos sobre la pared celular, en lugar de cubrirla homogéneamente como se podría esperar de una proteína que tiene un papel estabilizador (Figura 3). Entonces, ¿cómo es posible que las proteínas PIR estabilicen la pared si no la cubren totalmente? Recientemente, se observó que el β-1,3-glucano de la pared celular tiene una gran movilidad molecular; dado que las proteínas PIR se unen covalentemente a los β-1,3-glucanos, hipotetizamos que las proteínas PIR podrían ser estabilizadores locales de esos polisacáridos. Sin embargo, el mecanismo exacto de estabilización de estas extraordinarias, aunque discretas proteínas, serán el objetivo de investigaciones que están por venir.
Para saber más
Latgé, J.-P. & Wang, T. Modern Biophysics Redefines Our Understanding of Fungal Cell Wall Structure, Complexity, and Dynamics. mBio 13, e01145-22 (2022).
Urbar-Ulloa J, Montaño-Silva P, Ramírez-Pelayo, S et al. Cell surface display of proteins on filamentous fungi. Applied Microbiology and Biotechnology 103, 6949–6972 (2019).
Montaño-Silva, P., Callejas-Negrete, O. A., Pereira-Santana, A. & Verdin, J. Cell wall-resident proteins with internal repeats (PIRs) show an inverted architecture in Neurospora crassa, but maintain their role as wall stabilizers. FEBS Journal, doi: 10.1111/febs.70020 (2025).
1Unidad de Biotecnología Industrial, Centro de Investigación y Asistencia en Tecnología y Diseño del Estado de Jalisco
2Departamento de Microbiología, Centro de Investigación Científica y de Educación Superior de Ensenada
3SECIHTI-Centro de Investigación y Asistencia en Tecnología y Diseño del Estado de Jalisco, Sede Sureste

